들어가며
코로나-19(COVID-19) 사태로 대한민국에서 가장 바쁜신 분들이 바로 질병관리본부 직원들이지 않을까 싶습니다.
(국민의 한사람으로서 이 자리를 빌어 감사의 인사를 전합니다.)
오늘은 그 질병관리본부에서 관리하고 조사하는 '국민건강영양조사'라는 데이터를 소개해 볼까 합니다.
제목에서 느껴지시겠지만 건강과 영양섭취에 대한 우리나라 국가 통계입니다.
아래 최근 기사를 보시면 국민건강영양조사를 통해 코로나19의 면역여부를 체크해보겠다는 내용같습니다.
코로나19 집단면역 조사한다... 당국 "국민영양조사 통해 확인" - 연합뉴스 2020-04-30
코로나19 집단면역 조사한다…당국 "국민영양조사 통해 확인"(종합) | 연합뉴스
코로나19 집단면역 조사한다…당국 "국민영양조사 통해 확인"(종합), 신선미기자, 사회뉴스 (송고시간 2020-04-30 15:13)
www.yna.co.kr
그럼 국민건강영양조사는 어떤 식으로 이루어 질까요?
국민건강영양조사 홈페이지에서 자세히 안내가 되어 있습니다만 간단이 설명하자면 전문조사수행팀이 매주 4개지역 전국적으로 192개 지역을 방문하여 이동검진차량을 통해 방문검진과 건강설문조사를 하는 방식입니다.
전국의 192개 지역과 조사 대상은 대한민국의 인구, 가구 특성을 반영할수 있는 대표 가구를 별도로 샘플링 하는 것으로 알고 있습니다. 제가 살던 부산지역은 예전에 남구 용호동에 LG메트로시티 아파트에 매년 왔던것으로 기억합니다.
이동 검진차량으로 건강검진을 겸해서 수행하고, 나머지는 설문조사를 통해서 영양소, 식품 섭취량 조사를 하는 방식입니다.
데이터와 활용
해당 데이터는 국가주요통계로 대한민국 국민의 건강상태와 영양상태를 체크하고, 보건 정책 방향에도 많이 활용되는 자료입니다. 대학이나 병원등의 연구기관에서는 식품영양학, 간호학, 의학쪽에서 관련 자료를 이용해 연구하고 관련 논문도 많이 생산되고 있습니다.
각설하고 이제 그 데이터를 한번 알아보기로 하겠습니다.
데이터는 질본산하의 국민건강영양조사 홈페이지를 통해서 직접 다운 받을 수 있습니다.
 |
 |
원시자료 제공은 SAS와 SPSS용으로만 제공되고 있고 데이터셋 파일은 기본DB/ 검진조사/ 영양조사 이렇게 3종류입니다.
데이터 탐색
원시자료의 자세한 내용과 변수 및 설문영역에 대한 내용은 함께 제공되는 원시자료 이용지침서를 참고하시면 됩니다.
(무려 312페이지에 달하는 책자라서 처음에는 엄두가 안날수 도 있습니다.)
이용지치섬에서 보면 연도별로 조사영역과 조사에 해당하는 파일, 자료수와 변수의 갯수가 함께 제공됩니다.
제 7기의 세번째인 2018년기준으로 보면 가구조사/건강면접조사/영양조사/검진조사기본은 기본DB(Hn18_all)에 포함되어 있고, 검진조사의 하부 항목인 구강검사부분을 7기 전체로 해서 조사한 구강검사DB(HNY7_oe)가 있고, 마지막으로 식품섭취조사(hn18_24rc)DB 이렇게 총 3가지 파일로 제공되고 있습니다.
식이보충제 조사도 7기 2018년에 한걸로 되어 있는데 원시자료 다운로드 화면에서 별도 제공이 안된는 것으로 보이네요
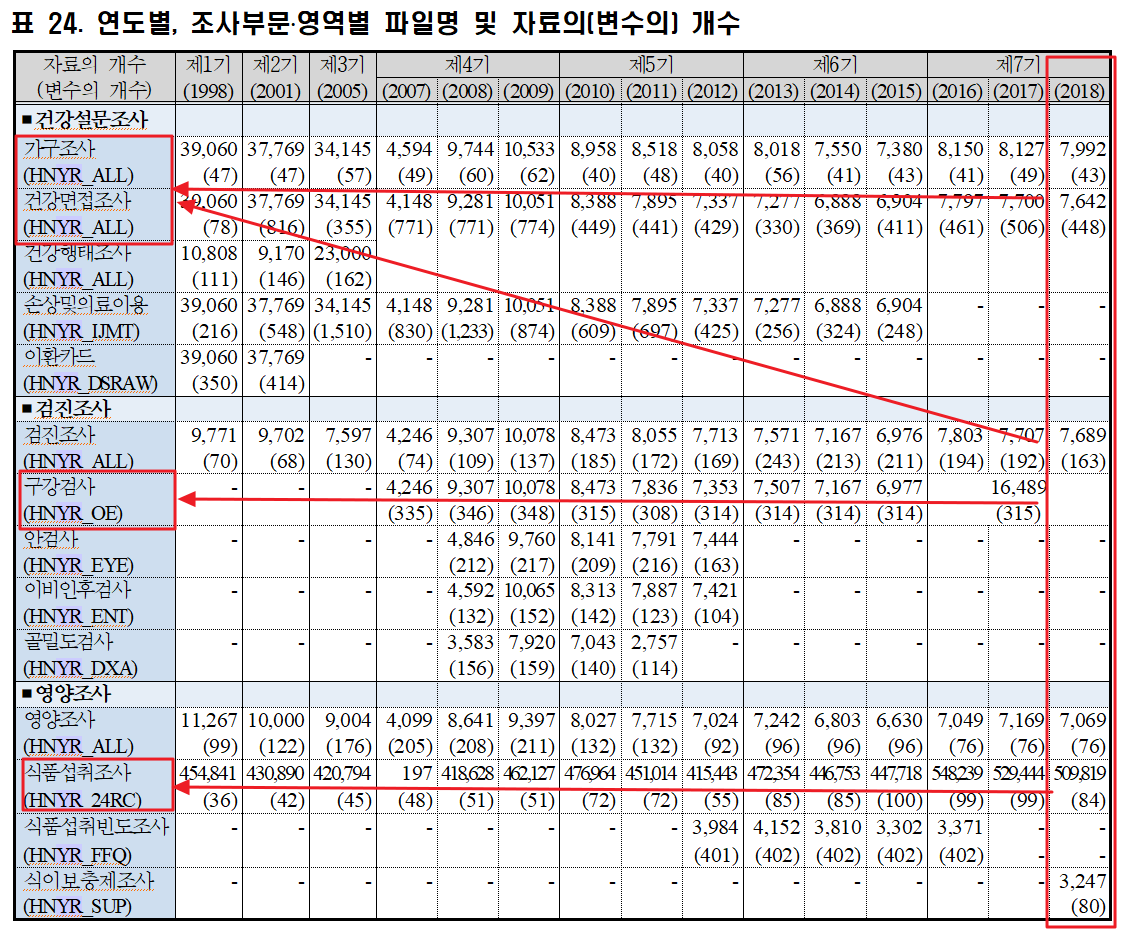
이제부터는 다운받은 파일을 가지고 직접 데이터 구조와 변수에 대한 내용을 알아보겠습니다. 2018년도 데이터셋 3개중에서 검진조사에 들어가 구강검사(HNY7_oe)부분은 제외하고 기본DB(Hn18_all)와 식품섭취조사(hn18_24rc)데이터셋을 집중적으로 알아보겠습니다.
저는 SPSS용 파일(확장자 sav)파일을 받았습니다.
압축을 풀어보니 기본DB는 155 MB, 영양조사DB는 1.34GB 로 용량차이가 심하게 차이가 나고 생각보다 데이터량이 많습니다. 데이터파일인데 1G가 넘는다는건 데이터수가 많거나 변수 수가 상당히 많다는 의미인데. 식품섭취조사 파일의경우 데이터수가 50만개가 넘고 변수의경우는 84개 정도로 되어 있네요.
SPSS프로그램에서 데이터를 불러와서 확인해보겠습니다.
기본DB(Hn18_all.sav)
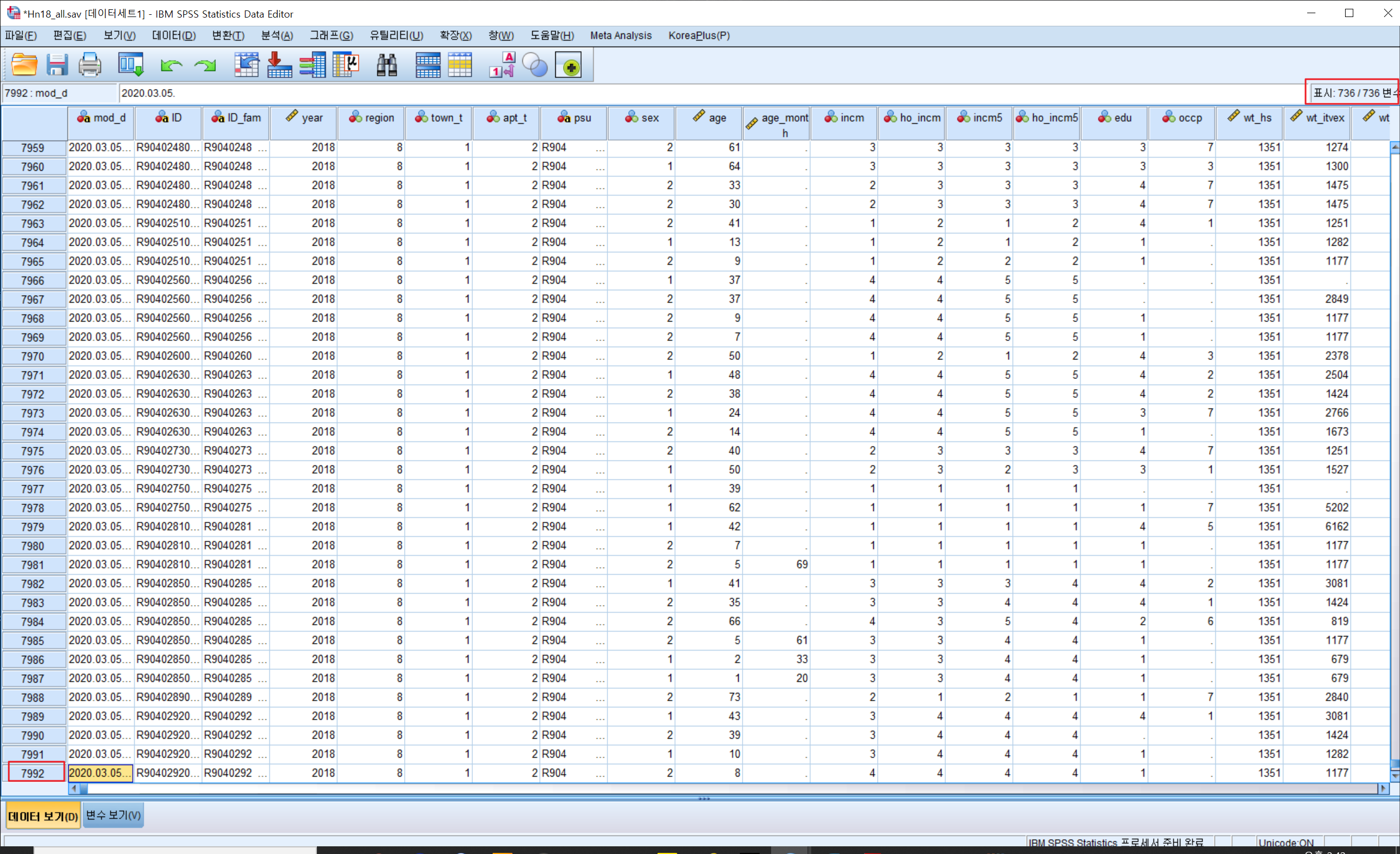
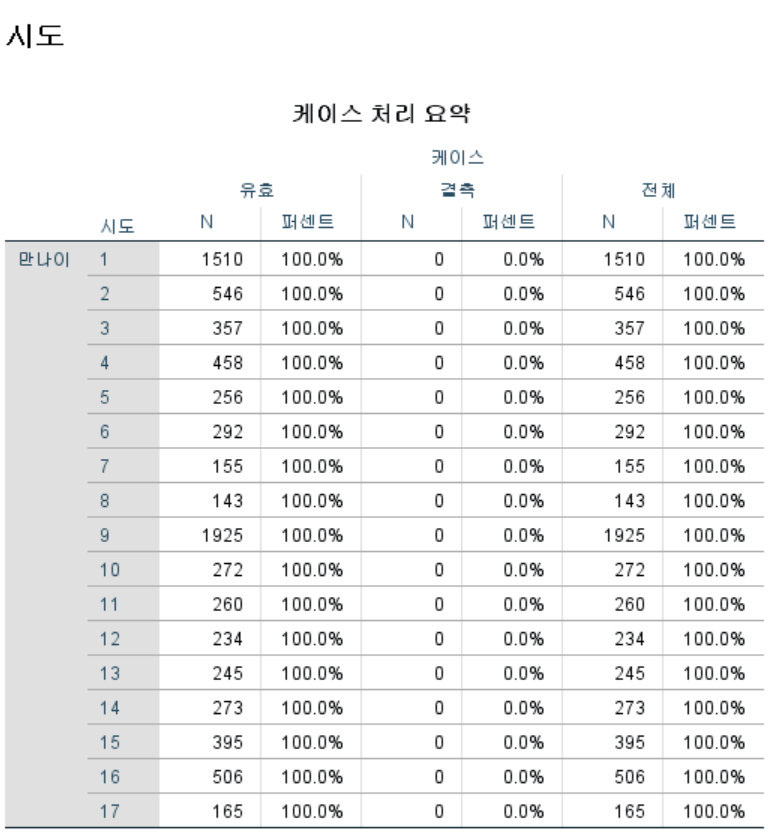
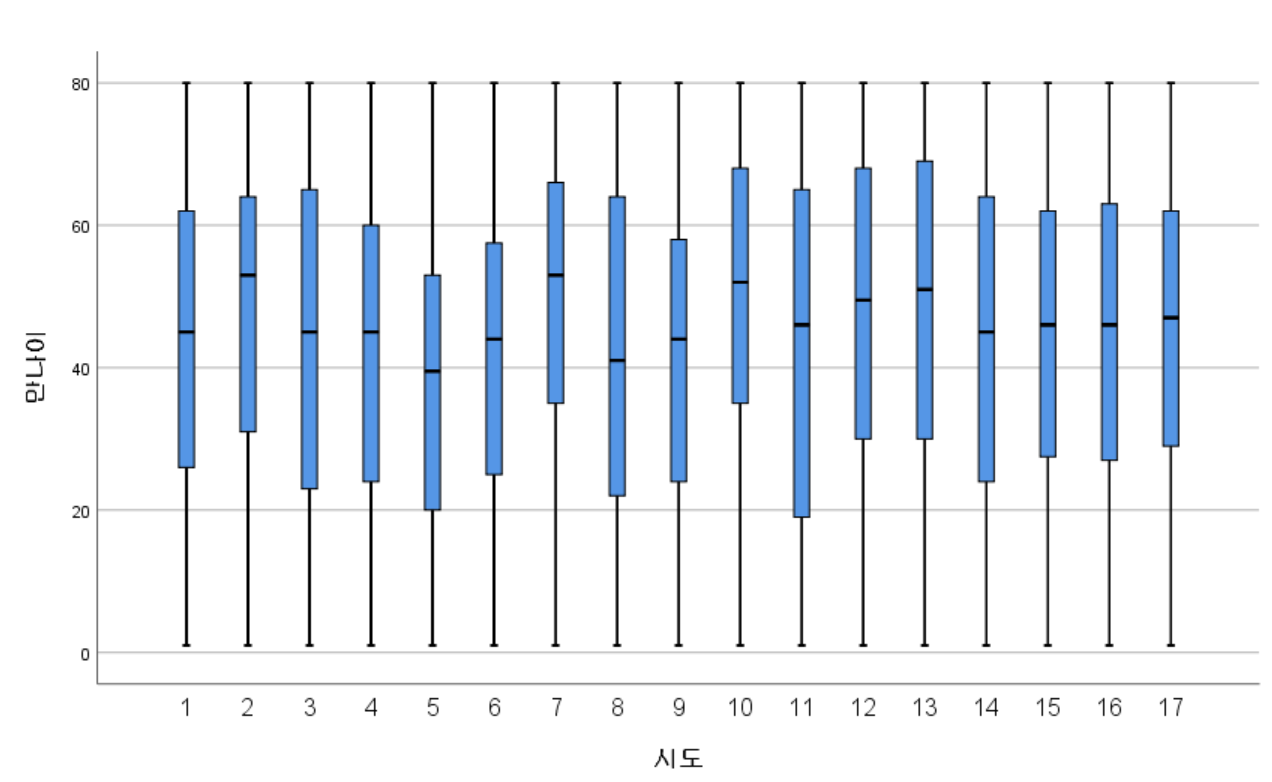
기본DB는 총 데이터수는 7,992개 변수의 수는 736개입니다. 기본DB에 가구조사, 건강조사, 영양조사 관련된 변수가 모두 포함되어 있어서 데이터수에 비해서 변수가 꽤 많은 편입니다. 간단이 지역별(region)로 나이별 분포로 샘플수를 체크해보았습니다. 아래 표를 보시면 17개 지역별로 샘플수가 1번인경우(아마 서울) 1510개 2번이 546개 이런식으로 나뉘어져있네요. 옆에 그립은 상자그림으로 만나이(age)의 분포를 지역별로 표시한 내용입니다.
 |
 |
코드들에 대한 자세한 내용은 이용지침서의 변수 설명 부분을 참고 하시면 됩니다.
지역의 경우는 이렇게 코드가 되어 있네요.
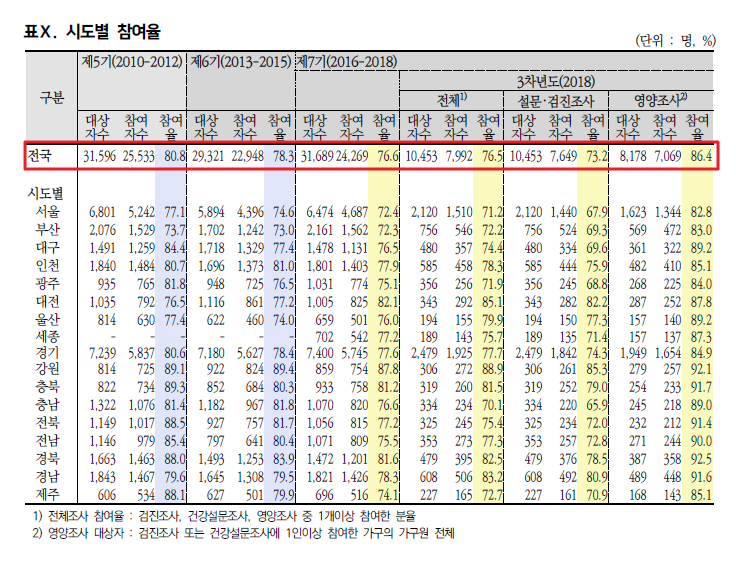
함께 받을 수 있는 자료인 2018국민건강통계 보고서에서 참여현황을 한번 보셔도 데이터 내용 파악하는데 도움이 될것 같습니다. 아래 표를 보시면 전국 기준으로 10453명의 대상자수에 실제 참여자수가 7992명으로 이 데이터가 기본데이터셋의 ID기준으로 실제 샘플수가 되는것 같습니다.
식품섭취조사DB(hn18_24rc.sav)
식품섭취조사데이터를 SPSS에서 읽어서 간단이 살펴보겠습니다.
해당데이터는 509819(열) * 114(변수)로 되어 있습니다.
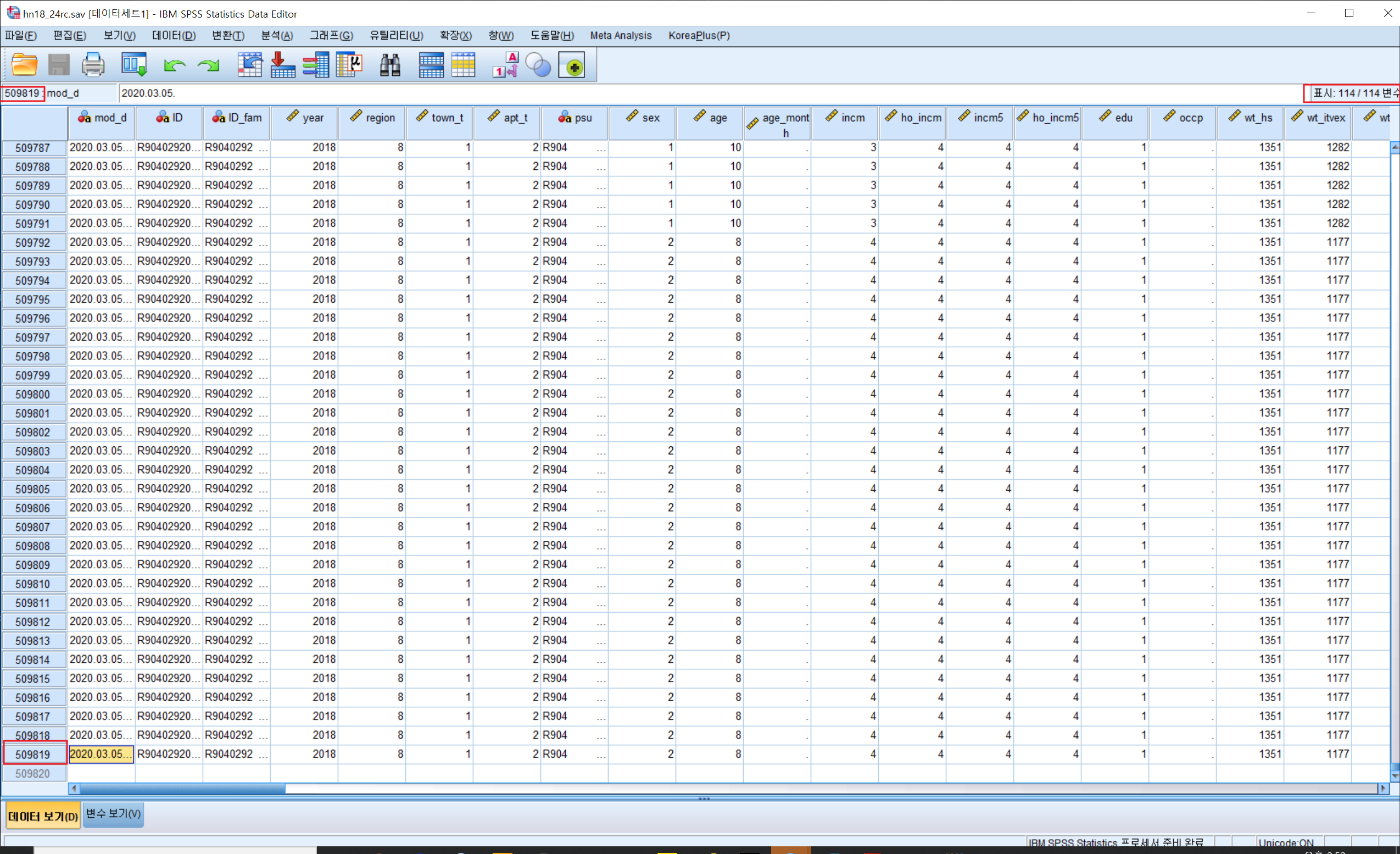
해당 데이터는 기본DB에 비해 ID기준으로 중복데이터가 많아보입니다. 이 부분은 ID기준(인당)으로 여러개의 데이터가 있다는 의미인데 자세히 보면 끼니별, 음식별로 데이터가 40여개에서 100여개까지 있습니다.
결국 ID기준으로보면 영양조사 참여자수인 7069명이 나와야 겠지요? 이부분은 나중에 다시 확인해보겠습니다.
SPSS형태에서도 기본적으로 많은 분석을 할 수 있지만, 전 R과 친해서 해당 파일을 R에서 읽어와서 탐색적데이터분석(EDA)을 한번 수행해서 데이터셋을 좀더 면밀이 살펴보겠습니다.
R로 데이터 살펴보기
제공되는 SPSS 파일은 확장자가 sav인데 실제 바이너리 파일 형태라 용량도 큰편입니다.
R에서 SPSS파일을 읽는법은 foreign 이나 haven같은 패키지를 이용해서 바로 읽는 방법이 있습니다만,
해당 데이터셋 같은 경우 파일의 인코딩 부분이 가끔 충돌을 일으키고 해서 저는 csv로 변환후 다시 R에서 읽는 방식이 편해서 그렇게 하고 있습니다. csv저장시에도 인코딩옵션을 UTF-8 또는 로컬 설정(EUC-KR)을 하시고 읽을때 해당 인코딩 옵션을 동일하게 해줘야 에러 없이 잘읽힙니다.
속도면에서도 csv변환후 R에서 읽어오는게(read.table) 훨씬 빠른 것 같습니다.
제가 자주 사용하는 코드는 아래와 같습니다. (저는 EUC-KR 로컬 인코딩으로 CSV저장후 처리했습니다)
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
|
library(readr)
read.csv.any <- function(text, sep = "", ...) {
encoding <- as.character(guess_encoding(text)[1,1])
setting <- as.character(tools::file_ext(text))
if(sep != "" | !(setting %in% c("csv", "txt")) ) setting <- "custom"
separate <- list(csv = ",", txt = "\n", custom = sep)
result <- read.table(text, sep = separate[[setting]], fileEncoding = encoding, ...)
return(result)
}
#기본db 읽어오기 : spss에서 csv저장시 encoding 로컬로 설정했음 이경우 EUC-KR
guess_encoding("./Hn18_all(spss)/Hn18_all_local.csv")
df_hn18_all <- read.csv("./Hn18_all(spss)/Hn18_all_local.csv", header = TRUE, sep = ",",fileEncoding = "EUC-KR")
#식품섭취조사db 읽어오기 : spss에서 csv저장시 encoding 로컬로 설정했음 이경우 EUC-KR
guess_encoding("./hn18_24rc(spss)/hn18_24rc_local.csv")
df_hn18_24rc <- read.csv("./hn18_24rc(spss)/hn18_24rc_local.csv", header = TRUE, sep = ",",fileEncoding = "EUC-KR")
|
cs |
R에서 데이터 프레임으로 변환후에 탐색적 데이터 분석을 DataExplore 란 패키지를 이용해서 한번 해보겠습니다.
기본DB(df_hn18_all)
| library("DataExplorer") introduce(df_hn18_all) rows columns discrete_columns continuous_columns all_missing_columns 7992 736 34 695 7 total_missing_values complete_rows total_observations memory_usage 1031466 0 5882112 27028720 |
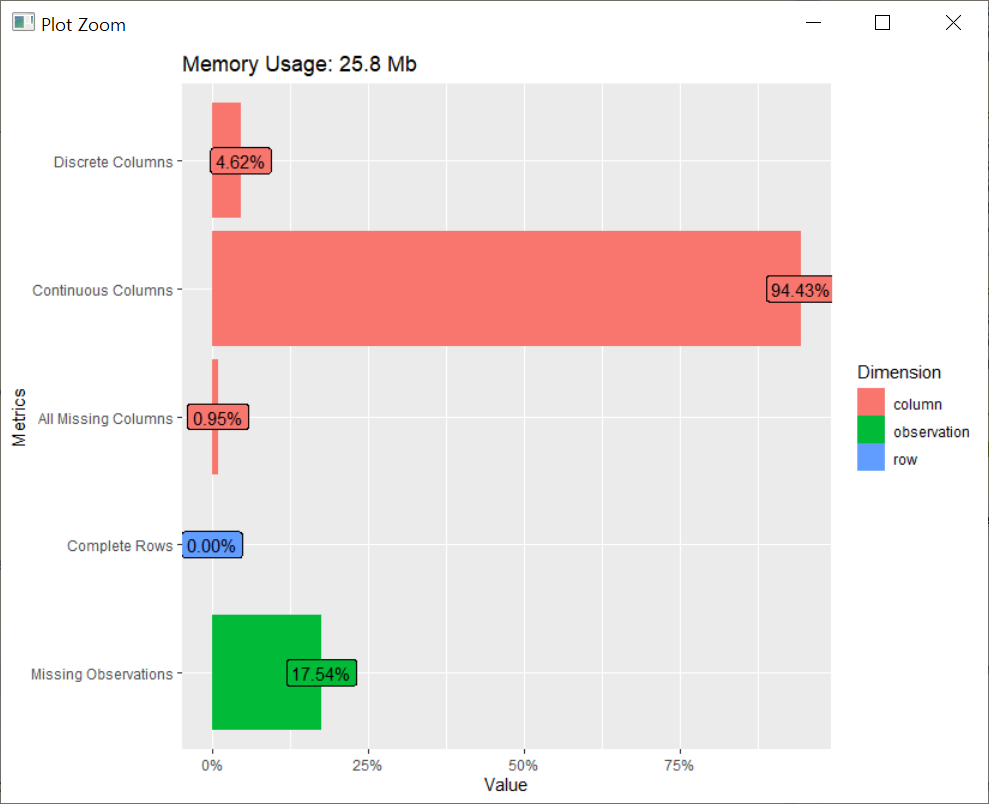
기본적인 소개(introduce)명령으로 데이터크기(rows) 7992, 변수(columns) 736/ 이산형데이터 컬럼(discrete_columns) 34/ 연속형 컬럼(continuous_columns) 695/ 전체결측컬럼(all_missing_columns)/결측데이터 수(total_missing_values)/ 메모리사용량(memory_usage)까지가 표시가 됩니다.
기본DB파일은 데이터수에 비해서 실제 변수가 736개로 많은편입니다.
이걸 plot_intro 명령으로 시각화해보면 아래처럼 확인이 가능합니다.
기본적으로 데이터 분석을 할때 결측치를 체크하는게 중요합니다.
plot_missing 펑션으로 한번 확인해보겠습니다. 변수가 너무 많아서(700여개) 결측치가 많은 변수가 하단에 표시가 되는데 쉽게 파악하기는 어렵습니다.
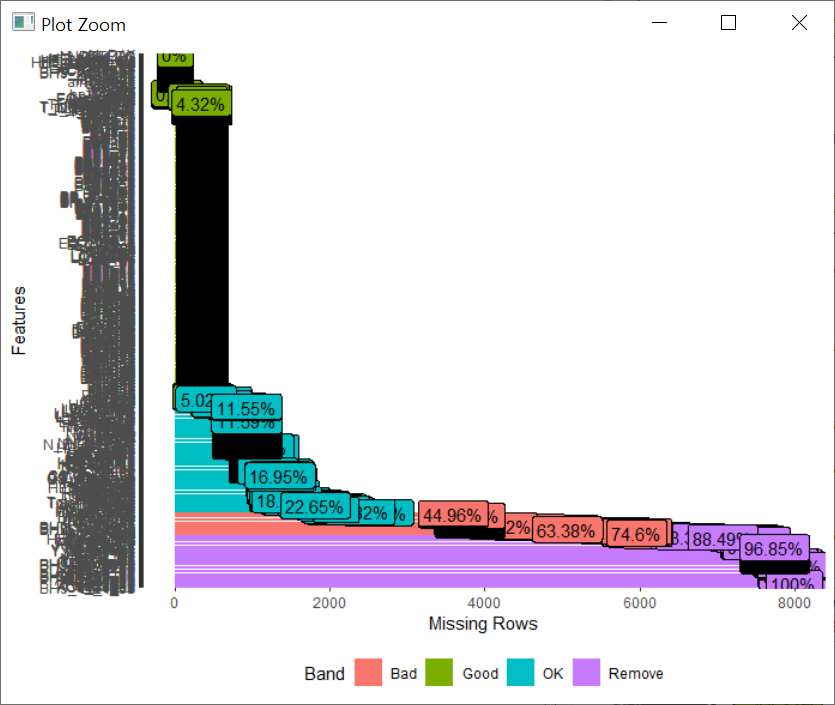
이 경우는 profile_missing 펑션을 이용해서 실제 컬럼별로 결측치 수와 비율 파악이 가능합니다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
|
df_hn18_all_missing <- profile_missing(df_hn18_all)
df_hn18_all_missing %>% filter(pct_missing ==1)
df_hn18_all_missing %>% filter(pct_missing >0.8) %>% arrange(desc(num_missing))
feature num_missing pct_missing
1 DE1_35 7992 1.0000000
2 BH9_14_4_03 7992 1.0000000
3 AC8_1e_02 7992 1.0000000
4 AC3_3e_03 7992 1.0000000
5 AC8_1e_03 7992 1.0000000
6 BS12_45 7992 1.0000000
7 BM13_6 7992 1.0000000
8 AC3_1_03 7991 0.9998749
9 AC3_2_03 7991 0.9998749
10 AC3_3_03 7991 0.9998749
11 AC8_1_03 7991 0.9998749
12 AC3_4_03 7991 0.9998749
13 AC8_2w_03 7991 0.9998749
14 AC8_2_03 7991 0.9998749
15 AC8_3w_03 7991 0.9998749
16 AC8_3_03 7991 0.9998749
17 BH9_14_1_03 7990 0.9997497
18 BH9_14_2_03 7990 0.9997497
19 BH9_14_3_03 7990 0.9997497
|
cs |
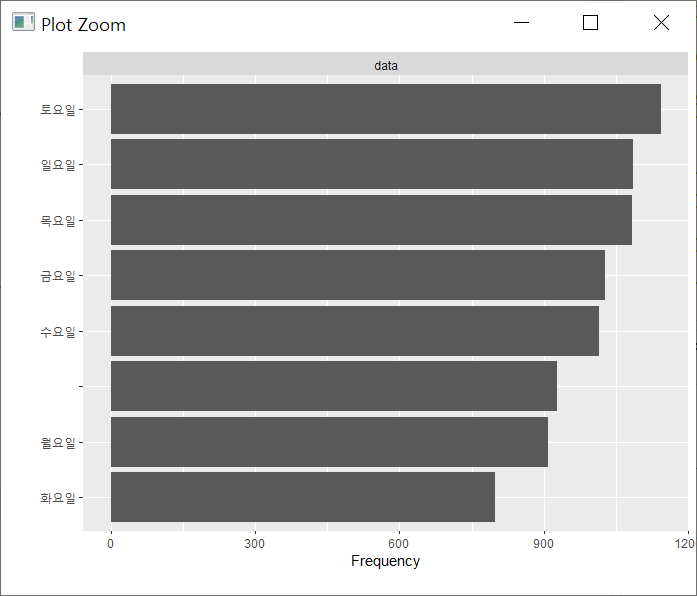
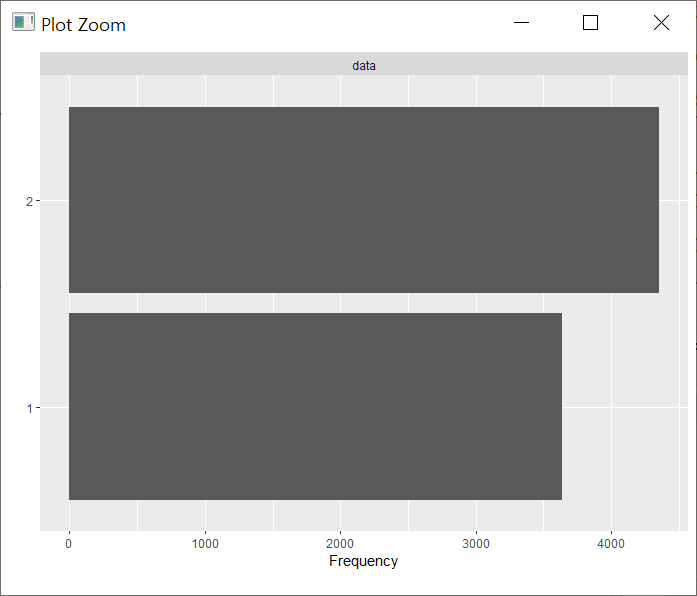
plot_bar를 통해서는 이산형 컬럼의 값 분포를 확인해볼수 있습니다.
|
1
2
3
4
|
#To visualize frequency distributions for all discrete features
plot_bar(df_hn18_all)
plot_bar(df_hn18_all$N_DAY)
plot_bar(df_hn18_all$sex)
|
cs |
 |
 |
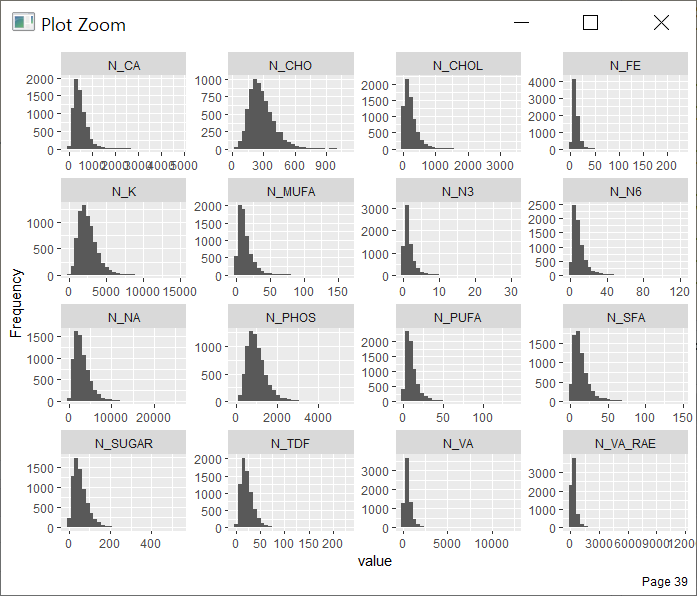
plot_histogram 이란 명령으로는 연속형 변수들의 분포를 확인해볼수 있습니다. 기본DB의 경우 변수의 수가 많아서 실제 수행시간이 약간 걸립니다. 변수별로 각각 1개씩 살펴볼수도 있습니다.
변수별로 정규성 검정을 하기위해서 QQ플롯(plot_qq)도 일괄적으로 확인해 볼 수 있습니다.
|
1
2
3
4
|
#QQ Plot
qq_data <- df_hn18_all[, c("age", "HE_ht", "HE_wt","N_INTK","N_EN","N_WATER")]
plot_qq(qq_data, sampled_rows = 1000L)
|
cs |

그 밖에도 상관관계(plot_correlation), 산점도(plot_scatterplot), 상자그림(plot_boxplot)등을 그려서 일괄로 시각화를 해서 살펴보는것도 가능합니다.
오늘은 변수(컬럼)가 많은 기본DB를 통해서 알아봤구요. 다음 포스팅에서는 데이터량이 많은 식품섭취조사 데이터를 가지고 추가적으로 탐색적 데이터분석(EDA)을 해보겠습니다.
함께 읽으면 좋을 글
국민건강영양조사
국민건강영양조사란? 국민건강영양조사는 국민건강증진법에 따라 매년 우리나라 국민 1만명에 대한 ...
blog.naver.com
국민건강영양조사 20년(1998~2018)
고기는 OK 운동은 NO 건강마저‘부익부 빈익빈’우리 국민, 20년간 흡연은 줄고 비만은 늘었다- 지난 2...
blog.naver.com
'아재도 하는 데이터분석' 카테고리의 다른 글
| [데이터소개] 스타벅스 매장은 지난 1년간 얼마나 늘었을까?(feat. 스타벅스 매장 목록 파일, 2023.06.15.) (1) | 2023.06.17 |
|---|---|
| [데이터 소개] 국민건강영양조사 2편(Feat. R DataExplorer) (1) | 2020.05.21 |
| [데이터분석] 한우 사육현황 데이터 시각화 feat by ggplot2 & Excel (2) | 2020.05.16 |
| [데이터분석] 일자리가 가장 많은 동네는?(feat by 국세통계 연말정산 신고현황) (0) | 2020.03.11 |
| [데이터분석] 파리바게뜨로 보는 부산지역 상권현황 (2) | 2020.03.09 |
들어가며
코로나-19(COVID-19) 사태로 대한민국에서 가장 바쁜신 분들이 바로 질병관리본부 직원들이지 않을까 싶습니다.
(국민의 한사람으로서 이 자리를 빌어 감사의 인사를 전합니다.)
오늘은 그 질병관리본부에서 관리하고 조사하는 '국민건강영양조사'라는 데이터를 소개해 볼까 합니다.
제목에서 느껴지시겠지만 건강과 영양섭취에 대한 우리나라 국가 통계입니다.
아래 최근 기사를 보시면 국민건강영양조사를 통해 코로나19의 면역여부를 체크해보겠다는 내용같습니다.
코로나19 집단면역 조사한다... 당국 "국민영양조사 통해 확인" - 연합뉴스 2020-04-30
코로나19 집단면역 조사한다…당국 "국민영양조사 통해 확인"(종합) | 연합뉴스
코로나19 집단면역 조사한다…당국 "국민영양조사 통해 확인"(종합), 신선미기자, 사회뉴스 (송고시간 2020-04-30 15:13)
www.yna.co.kr
그럼 국민건강영양조사는 어떤 식으로 이루어 질까요?
국민건강영양조사 홈페이지에서 자세히 안내가 되어 있습니다만 간단이 설명하자면 전문조사수행팀이 매주 4개지역 전국적으로 192개 지역을 방문하여 이동검진차량을 통해 방문검진과 건강설문조사를 하는 방식입니다.
전국의 192개 지역과 조사 대상은 대한민국의 인구, 가구 특성을 반영할수 있는 대표 가구를 별도로 샘플링 하는 것으로 알고 있습니다. 제가 살던 부산지역은 예전에 남구 용호동에 LG메트로시티 아파트에 매년 왔던것으로 기억합니다.
이동 검진차량으로 건강검진을 겸해서 수행하고, 나머지는 설문조사를 통해서 영양소, 식품 섭취량 조사를 하는 방식입니다.
데이터와 활용
해당 데이터는 국가주요통계로 대한민국 국민의 건강상태와 영양상태를 체크하고, 보건 정책 방향에도 많이 활용되는 자료입니다. 대학이나 병원등의 연구기관에서는 식품영양학, 간호학, 의학쪽에서 관련 자료를 이용해 연구하고 관련 논문도 많이 생산되고 있습니다.
각설하고 이제 그 데이터를 한번 알아보기로 하겠습니다.
데이터는 질본산하의 국민건강영양조사 홈페이지를 통해서 직접 다운 받을 수 있습니다.
 |
 |
원시자료 제공은 SAS와 SPSS용으로만 제공되고 있고 데이터셋 파일은 기본DB/ 검진조사/ 영양조사 이렇게 3종류입니다.
데이터 탐색
원시자료의 자세한 내용과 변수 및 설문영역에 대한 내용은 함께 제공되는 원시자료 이용지침서를 참고하시면 됩니다.
(무려 312페이지에 달하는 책자라서 처음에는 엄두가 안날수 도 있습니다.)
이용지치섬에서 보면 연도별로 조사영역과 조사에 해당하는 파일, 자료수와 변수의 갯수가 함께 제공됩니다.
제 7기의 세번째인 2018년기준으로 보면 가구조사/건강면접조사/영양조사/검진조사기본은 기본DB(Hn18_all)에 포함되어 있고, 검진조사의 하부 항목인 구강검사부분을 7기 전체로 해서 조사한 구강검사DB(HNY7_oe)가 있고, 마지막으로 식품섭취조사(hn18_24rc)DB 이렇게 총 3가지 파일로 제공되고 있습니다.
식이보충제 조사도 7기 2018년에 한걸로 되어 있는데 원시자료 다운로드 화면에서 별도 제공이 안된는 것으로 보이네요

이제부터는 다운받은 파일을 가지고 직접 데이터 구조와 변수에 대한 내용을 알아보겠습니다. 2018년도 데이터셋 3개중에서 검진조사에 들어가 구강검사(HNY7_oe)부분은 제외하고 기본DB(Hn18_all)와 식품섭취조사(hn18_24rc)데이터셋을 집중적으로 알아보겠습니다.
저는 SPSS용 파일(확장자 sav)파일을 받았습니다.
압축을 풀어보니 기본DB는 155 MB, 영양조사DB는 1.34GB 로 용량차이가 심하게 차이가 나고 생각보다 데이터량이 많습니다. 데이터파일인데 1G가 넘는다는건 데이터수가 많거나 변수 수가 상당히 많다는 의미인데. 식품섭취조사 파일의경우 데이터수가 50만개가 넘고 변수의경우는 84개 정도로 되어 있네요.
SPSS프로그램에서 데이터를 불러와서 확인해보겠습니다.
기본DB(Hn18_all.sav)

기본DB는 총 데이터수는 7,992개 변수의 수는 736개입니다. 기본DB에 가구조사, 건강조사, 영양조사 관련된 변수가 모두 포함되어 있어서 데이터수에 비해서 변수가 꽤 많은 편입니다. 간단이 지역별(region)로 나이별 분포로 샘플수를 체크해보았습니다. 아래 표를 보시면 17개 지역별로 샘플수가 1번인경우(아마 서울) 1510개 2번이 546개 이런식으로 나뉘어져있네요. 옆에 그립은 상자그림으로 만나이(age)의 분포를 지역별로 표시한 내용입니다.
 |
 |
코드들에 대한 자세한 내용은 이용지침서의 변수 설명 부분을 참고 하시면 됩니다.
지역의 경우는 이렇게 코드가 되어 있네요.

함께 받을 수 있는 자료인 2018국민건강통계 보고서에서 참여현황을 한번 보셔도 데이터 내용 파악하는데 도움이 될것 같습니다. 아래 표를 보시면 전국 기준으로 10453명의 대상자수에 실제 참여자수가 7992명으로 이 데이터가 기본데이터셋의 ID기준으로 실제 샘플수가 되는것 같습니다.

식품섭취조사DB(hn18_24rc.sav)
식품섭취조사데이터를 SPSS에서 읽어서 간단이 살펴보겠습니다.
해당데이터는 509819(열) * 114(변수)로 되어 있습니다.

해당 데이터는 기본DB에 비해 ID기준으로 중복데이터가 많아보입니다. 이 부분은 ID기준(인당)으로 여러개의 데이터가 있다는 의미인데 자세히 보면 끼니별, 음식별로 데이터가 40여개에서 100여개까지 있습니다.
결국 ID기준으로보면 영양조사 참여자수인 7069명이 나와야 겠지요? 이부분은 나중에 다시 확인해보겠습니다.
SPSS형태에서도 기본적으로 많은 분석을 할 수 있지만, 전 R과 친해서 해당 파일을 R에서 읽어와서 탐색적데이터분석(EDA)을 한번 수행해서 데이터셋을 좀더 면밀이 살펴보겠습니다.
R로 데이터 살펴보기
제공되는 SPSS 파일은 확장자가 sav인데 실제 바이너리 파일 형태라 용량도 큰편입니다.
R에서 SPSS파일을 읽는법은 foreign 이나 haven같은 패키지를 이용해서 바로 읽는 방법이 있습니다만,
해당 데이터셋 같은 경우 파일의 인코딩 부분이 가끔 충돌을 일으키고 해서 저는 csv로 변환후 다시 R에서 읽는 방식이 편해서 그렇게 하고 있습니다. csv저장시에도 인코딩옵션을 UTF-8 또는 로컬 설정(EUC-KR)을 하시고 읽을때 해당 인코딩 옵션을 동일하게 해줘야 에러 없이 잘읽힙니다.
속도면에서도 csv변환후 R에서 읽어오는게(read.table) 훨씬 빠른 것 같습니다.
제가 자주 사용하는 코드는 아래와 같습니다. (저는 EUC-KR 로컬 인코딩으로 CSV저장후 처리했습니다)
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
|
library(readr)
read.csv.any <- function(text, sep = "", ...) {
encoding <- as.character(guess_encoding(text)[1,1])
setting <- as.character(tools::file_ext(text))
if(sep != "" | !(setting %in% c("csv", "txt")) ) setting <- "custom"
separate <- list(csv = ",", txt = "\n", custom = sep)
result <- read.table(text, sep = separate[[setting]], fileEncoding = encoding, ...)
return(result)
}
#기본db 읽어오기 : spss에서 csv저장시 encoding 로컬로 설정했음 이경우 EUC-KR
guess_encoding("./Hn18_all(spss)/Hn18_all_local.csv")
df_hn18_all <- read.csv("./Hn18_all(spss)/Hn18_all_local.csv", header = TRUE, sep = ",",fileEncoding = "EUC-KR")
#식품섭취조사db 읽어오기 : spss에서 csv저장시 encoding 로컬로 설정했음 이경우 EUC-KR
guess_encoding("./hn18_24rc(spss)/hn18_24rc_local.csv")
df_hn18_24rc <- read.csv("./hn18_24rc(spss)/hn18_24rc_local.csv", header = TRUE, sep = ",",fileEncoding = "EUC-KR")
|
cs |
R에서 데이터 프레임으로 변환후에 탐색적 데이터 분석을 DataExplore 란 패키지를 이용해서 한번 해보겠습니다.
기본DB(df_hn18_all)
| library("DataExplorer") introduce(df_hn18_all) rows columns discrete_columns continuous_columns all_missing_columns 7992 736 34 695 7 total_missing_values complete_rows total_observations memory_usage 1031466 0 5882112 27028720 |
기본적인 소개(introduce)명령으로 데이터크기(rows) 7992, 변수(columns) 736/ 이산형데이터 컬럼(discrete_columns) 34/ 연속형 컬럼(continuous_columns) 695/ 전체결측컬럼(all_missing_columns)/결측데이터 수(total_missing_values)/ 메모리사용량(memory_usage)까지가 표시가 됩니다.
기본DB파일은 데이터수에 비해서 실제 변수가 736개로 많은편입니다.
이걸 plot_intro 명령으로 시각화해보면 아래처럼 확인이 가능합니다.

기본적으로 데이터 분석을 할때 결측치를 체크하는게 중요합니다.
plot_missing 펑션으로 한번 확인해보겠습니다. 변수가 너무 많아서(700여개) 결측치가 많은 변수가 하단에 표시가 되는데 쉽게 파악하기는 어렵습니다.

이 경우는 profile_missing 펑션을 이용해서 실제 컬럼별로 결측치 수와 비율 파악이 가능합니다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
|
df_hn18_all_missing <- profile_missing(df_hn18_all)
df_hn18_all_missing %>% filter(pct_missing ==1)
df_hn18_all_missing %>% filter(pct_missing >0.8) %>% arrange(desc(num_missing))
feature num_missing pct_missing
1 DE1_35 7992 1.0000000
2 BH9_14_4_03 7992 1.0000000
3 AC8_1e_02 7992 1.0000000
4 AC3_3e_03 7992 1.0000000
5 AC8_1e_03 7992 1.0000000
6 BS12_45 7992 1.0000000
7 BM13_6 7992 1.0000000
8 AC3_1_03 7991 0.9998749
9 AC3_2_03 7991 0.9998749
10 AC3_3_03 7991 0.9998749
11 AC8_1_03 7991 0.9998749
12 AC3_4_03 7991 0.9998749
13 AC8_2w_03 7991 0.9998749
14 AC8_2_03 7991 0.9998749
15 AC8_3w_03 7991 0.9998749
16 AC8_3_03 7991 0.9998749
17 BH9_14_1_03 7990 0.9997497
18 BH9_14_2_03 7990 0.9997497
19 BH9_14_3_03 7990 0.9997497
|
cs |
plot_bar를 통해서는 이산형 컬럼의 값 분포를 확인해볼수 있습니다.
|
1
2
3
4
|
#To visualize frequency distributions for all discrete features
plot_bar(df_hn18_all)
plot_bar(df_hn18_all$N_DAY)
plot_bar(df_hn18_all$sex)
|
cs |
 |
 |
plot_histogram 이란 명령으로는 연속형 변수들의 분포를 확인해볼수 있습니다. 기본DB의 경우 변수의 수가 많아서 실제 수행시간이 약간 걸립니다. 변수별로 각각 1개씩 살펴볼수도 있습니다.

변수별로 정규성 검정을 하기위해서 QQ플롯(plot_qq)도 일괄적으로 확인해 볼 수 있습니다.
|
1
2
3
4
|
#QQ Plot
qq_data <- df_hn18_all[, c("age", "HE_ht", "HE_wt","N_INTK","N_EN","N_WATER")]
plot_qq(qq_data, sampled_rows = 1000L)
|
cs |

그 밖에도 상관관계(plot_correlation), 산점도(plot_scatterplot), 상자그림(plot_boxplot)등을 그려서 일괄로 시각화를 해서 살펴보는것도 가능합니다.
오늘은 변수(컬럼)가 많은 기본DB를 통해서 알아봤구요. 다음 포스팅에서는 데이터량이 많은 식품섭취조사 데이터를 가지고 추가적으로 탐색적 데이터분석(EDA)을 해보겠습니다.
함께 읽으면 좋을 글
국민건강영양조사
국민건강영양조사란? 국민건강영양조사는 국민건강증진법에 따라 매년 우리나라 국민 1만명에 대한 ...
blog.naver.com
국민건강영양조사 20년(1998~2018)
고기는 OK 운동은 NO 건강마저‘부익부 빈익빈’우리 국민, 20년간 흡연은 줄고 비만은 늘었다- 지난 2...
blog.naver.com
'아재도 하는 데이터분석' 카테고리의 다른 글
| [데이터소개] 스타벅스 매장은 지난 1년간 얼마나 늘었을까?(feat. 스타벅스 매장 목록 파일, 2023.06.15.) (1) | 2023.06.17 |
|---|---|
| [데이터 소개] 국민건강영양조사 2편(Feat. R DataExplorer) (1) | 2020.05.21 |
| [데이터분석] 한우 사육현황 데이터 시각화 feat by ggplot2 & Excel (2) | 2020.05.16 |
| [데이터분석] 일자리가 가장 많은 동네는?(feat by 국세통계 연말정산 신고현황) (0) | 2020.03.11 |
| [데이터분석] 파리바게뜨로 보는 부산지역 상권현황 (2) | 2020.03.09 |